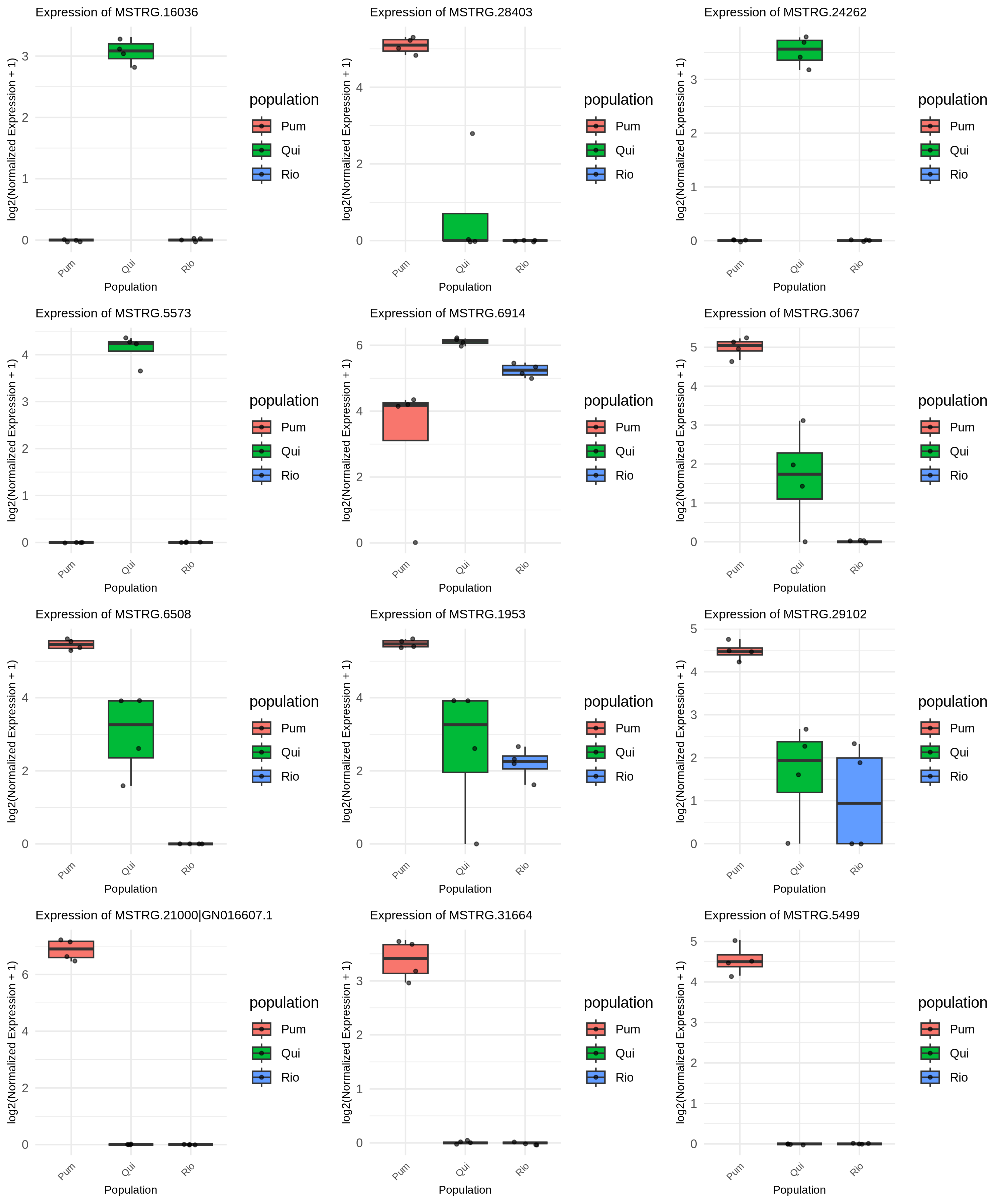
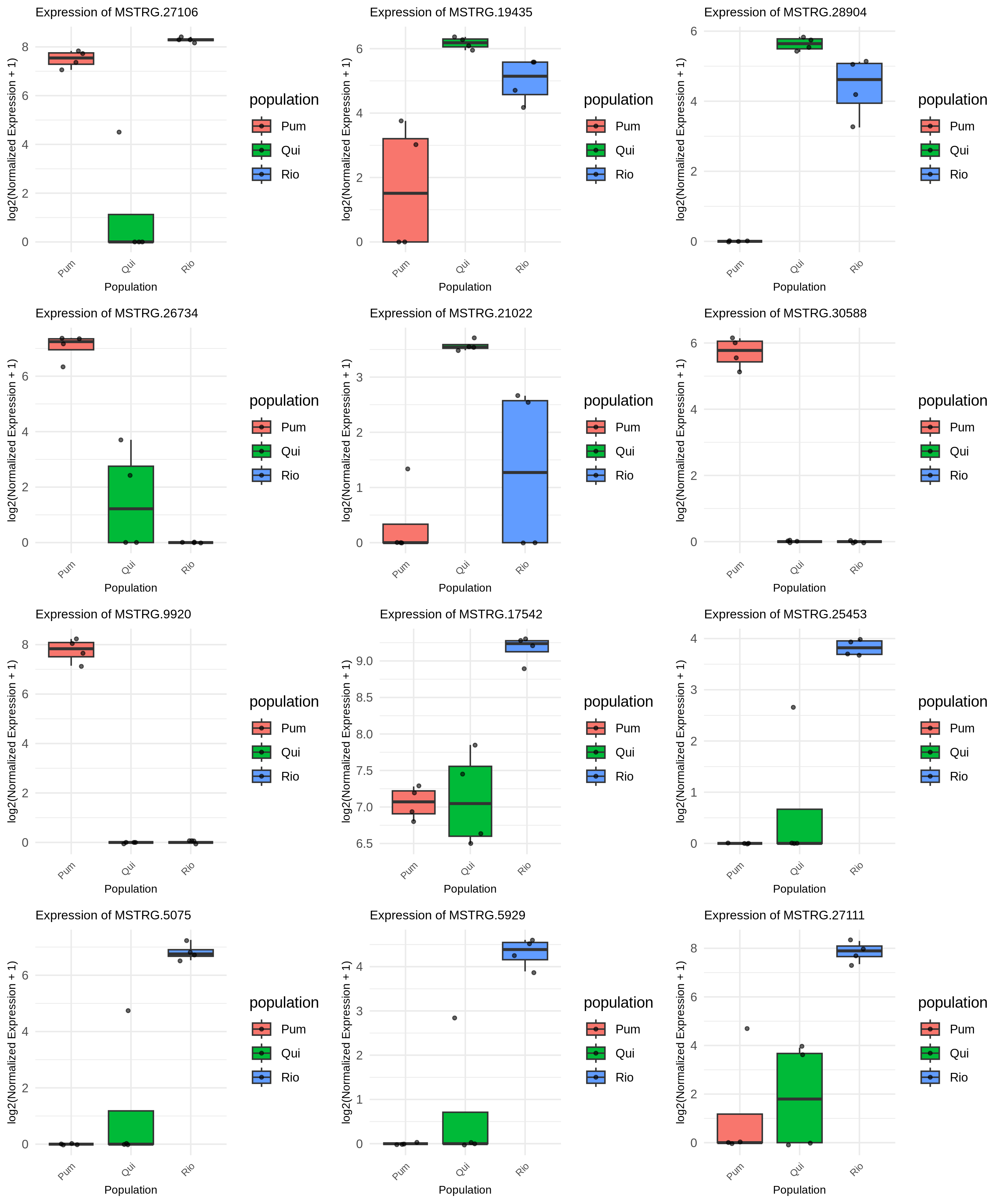
| gene_id | contrast | X1 | X2 | X3 | X4 | X5 | X6 | X7 | X8 | X9 | X10 | X11 | X12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| MSTRG.16036 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.28403 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.24262 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5573 | Pum_vs_Qui | MSTRG.5573::Chromosome_1B:170312406-170333998(+) | sp|Q5TZA2|CROCC_HUMAN | 48.0 | 98 | 48 | 2 | 4152 | 4439 | 22 | 118 | 0.0e+00 | 77.0 |
| MSTRG.5573 | Pum_vs_Qui | MSTRG.5573::Chromosome_1B:170312557-170362327(+) | sp|Q5TZA2|CROCC_HUMAN | 48.0 | 98 | 48 | 2 | 4001 | 4288 | 22 | 118 | 0.0e+00 | 77.0 |
| MSTRG.6914 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.3067 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.6508 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.1953 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.29102 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.21000|GN016607.1 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.31664 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5499 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.27106 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.19435 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.28904 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.26734 | Pum_vs_Qui | MSTRG.26734::Chromosome_7B:129126903-129127351(.) | sp|Q9BXX0|EMIL2_HUMAN | 40.5 | 74 | 42 | 1 | 431 | 210 | 906 | 977 | 4.5e-06 | 48.5 |
| MSTRG.21022 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.30588 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.9920 | Pum_vs_Qui | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.6508 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.28403 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.3067 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.1953 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.17542 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.29102 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.25453 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.21000|GN016607.1 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5075 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5929 | Pum_vs_Rio | MSTRG.5929::Chromosome_1B:186679521-186686006(+) | sp|Q6DIY8|M17L2_XENTR | 38.4 | 198 | 116 | 4 | 5682 | 6266 | 2 | 196 | 0.0e+00 | 124.0 |
| MSTRG.31664 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5499 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.27111 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.6115 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.26734 | Pum_vs_Rio | MSTRG.26734::Chromosome_7B:129126903-129127351(.) | sp|Q9BXX0|EMIL2_HUMAN | 40.5 | 74 | 42 | 1 | 431 | 210 | 906 | 977 | 4.5e-06 | 48.5 |
| MSTRG.31461 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.18807 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.30588 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.2111 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.9920 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.21438 | Pum_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.27106 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.16036 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.24262 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5573 | Qui_vs_Rio | MSTRG.5573::Chromosome_1B:170312406-170333998(+) | sp|Q5TZA2|CROCC_HUMAN | 48.0 | 98 | 48 | 2 | 4152 | 4439 | 22 | 118 | 0.0e+00 | 77.0 |
| MSTRG.5573 | Qui_vs_Rio | MSTRG.5573::Chromosome_1B:170312557-170362327(+) | sp|Q5TZA2|CROCC_HUMAN | 48.0 | 98 | 48 | 2 | 4001 | 4288 | 22 | 118 | 0.0e+00 | 77.0 |
| MSTRG.17542 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.25471 | Qui_vs_Rio | MSTRG.25471::Chromosome_7B:80510238-80537915(+) | sp|A9ULZ2|BIR7B_XENLA | 32.6 | 190 | 91 | 4 | 10080 | 10538 | 154 | 343 | 0.0e+00 | 112.0 |
| MSTRG.25471 | Qui_vs_Rio | MSTRG.25471::Chromosome_7B:80510238-80537915(+) | sp|A9ULZ2|BIR7B_XENLA | 32.6 | 190 | 91 | 4 | 10080 | 10538 | 154 | 343 | 0.0e+00 | 112.0 |
| MSTRG.24917 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.10366 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.25453 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.27117 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5075 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.27111 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.24906 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.5929 | Qui_vs_Rio | MSTRG.5929::Chromosome_1B:186679521-186686006(+) | sp|Q6DIY8|M17L2_XENTR | 38.4 | 198 | 116 | 4 | 5682 | 6266 | 2 | 196 | 0.0e+00 | 124.0 |
| MSTRG.6115 | Qui_vs_Rio | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| MSTRG.22169 | Qui_vs_Rio | MSTRG.22169::Chromosome_6B:76312513-76318400(-) | sp|Q8BFQ2|T229B_MOUSE | 60.9 | 161 | 57 | 1 | 5323 | 5787 | 3 | 163 | 0.0e+00 | 208.0 |
Started over with merged genome….
minimap2
eval "$(/opt/anaconda/anaconda3/bin/conda shell.bash hook)"
conda activate
# Set number of threads
threads=42
# Set paths
ref_genome="../data/merged_out.fasta.TBtools.fa"
input_dir="../output/02-RNAseq"
output_dir="../output/04-align-v1"
# Loop over each FASTQ file
for fq in ${input_dir}/*.fastq.gz; do
# Extract sample name (e.g., Pum_barcode02 from Pum_barcode02_combined.fastq.gz)
sample=$(basename "$fq" .fastq.gz)
echo "Processing $sample..."
# Run minimap2 and samtools sort
minimap2 -ax splice -k14 --MD -t $threads "$ref_genome" "$fq" | \
samtools sort -@ $threads -o "${output_dir}/${sample}.sorted.bam"
# Index the sorted BAM
samtools index "${output_dir}/${sample}.sorted.bam"
doneStringtie
mkdir -p ../output/04-align-v1/stringtie_out
for bam in ../output/04-align-v1/*combined.sorted.bam; do
sample=$(basename "$bam" _combined.sorted.bam)
/home/shared/stringtie-2.2.1.Linux_x86_64/stringtie "$bam" \
-L \
-o ../output/04-align-v1/stringtie_out/${sample}.gtf \
-p 32 \
-v
donels ../output/04-align-v1/stringtie_out/*.gtf > ../output/04-align-v1/stringtie_out/mergelist.txt
/home/shared/stringtie-2.2.1.Linux_x86_64/stringtie --merge \
-L \
-G ../data/GN.gene.gtf \
-o ../output/04-align-v1/stringtie_out/merged.gtf \
../output/04-align-v1/stringtie_out/mergelist.txtmkdir -p ../output/04-align-v1/stringtie_quant
for bam in ../output/04-align-v1/*combined.sorted.bam; do
sample=$(basename "$bam" _combined.sorted.bam)
/home/shared/stringtie-2.2.1.Linux_x86_64/stringtie "$bam" \
-L \
-e \
-B \
-G ../output/04-align-v1/stringtie_out/merged.gtf \
-o ../output/04-align-v1/stringtie_quant/${sample}.gtf
donematrix
python /home/shared/stringtie-2.2.1.Linux_x86_64/prepDE.py \
-i ../output/04-align-v1/stringtie_quant/samplelist.txt \
-g ../output/04-align-v1/stringtie_quant/gene_count_matrix.csv \
-t ../output/04-align-v1/stringtie_quant/transcript_count_matrix.csvnormalize matrix
library(DESeq2)
# Load raw counts
gene_counts <- read.csv("../output/04-align-v1/stringtie_quant/gene_count_matrix.csv", row.names = 1)
# Make sample metadata
sample_info <- data.frame(
row.names = colnames(gene_counts),
condition = c("A", "A", "A","A", "B", "B", "B","B","C","C", "C", "C") # example
)
# DESeq2 object and normalization
dds <- DESeqDataSetFromMatrix(countData = gene_counts, colData = sample_info, design = ~ condition)
dds <- DESeq(dds)
norm_counts <- counts(dds, normalized = TRUE)limma (differential expression)
library(limma)
library(tidyverse)
# Prepare expression matrix (genes x samples)
expr <- as.matrix(norm_counts)
# Create design matrix
group <- factor(case_when(
str_detect(colnames(expr), "^Pum") ~ "Pum",
str_detect(colnames(expr), "^Qui") ~ "Qui",
str_detect(colnames(expr), "^Rio") ~ "Rio"
))
design <- model.matrix(~ 0 + group)
colnames(design) <- levels(group)
# Fit linear model
fit <- lmFit(expr, design)
# Create contrast for all pairwise comparisons
contrast_matrix <- makeContrasts(
Pum_vs_Qui = Pum - Qui,
Pum_vs_Rio = Pum - Rio,
Qui_vs_Rio = Qui - Rio,
levels = design
)
fit2 <- contrasts.fit(fit, contrast_matrix)
fit2 <- eBayes(fit2)
# Get top DE genes across comparisons
top_table <- topTable(fit2, coef = "Qui_vs_Rio", number = 20)
head(top_table)Get all Contrast
# Get all contrast names
contrast_names <- colnames(fit2$contrasts)
# Extract all DEGs per contrast
deg_list <- lapply(contrast_names, function(contrast) {
tt <- topTable(fit2, coef = contrast, number = Inf, sort.by = "P") # all genes
tt$gene_id <- rownames(tt)
tt$contrast <- contrast
tt
})
# Combine into one long data frame
all_degs <- bind_rows(deg_list)sig_degs %>%
count(contrast)
library(UpSetR)
# Make DEG sets by contrast
deg_sets <- sig_degs %>%
group_by(contrast) %>%
summarize(genes = list(gene_id)) %>%
deframe()
upset(fromList(deg_sets), nsets = length(deg_sets), order.by = "freq")